
 媒体聚焦
媒体聚焦
DNA计算新突破!上海交大樊春海王飞团队开发支持通用性数字计算的DNA可编程门阵列
新民晚报
新民晚报讯(记者 易蓉)1994年,图灵奖得主Adleman提出利用DNA的碱基互补配对原则来发展生物计算,开启先河。但现有DNA计算体系仅能针对特定功能进行硬件定制,而电子计算机领域的如FPGA则可以通过软件编程的方式就能执行各种运算功能,无须从头设计制造硬件。因此,如何发展具有通用性的DNA运算元件的编程与集成已成为制约DNA计算领域发展的瓶颈。
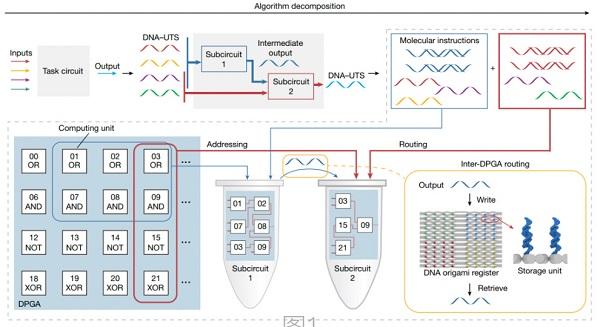
图说:实现器件内可编程性和器件间可集成性 采访对象供图(下同)
记者今天从上海交通大学获悉,上海交通大学化学化工学院/变革性分子前沿科学中心樊春海院士与王飞副教授近期发展了一种支持通用性数字计算的DNA可编程门阵列(DNA-based programmable gate array, DPGA),可通过分子指令编程的方式实现通用数字DNA计算,实现了无衰减大规模液相分子电路的构建。该成果近日发表于《自然》(Nature)杂志。
基于DNA分子间相互作用的液相DNA分子计算在高并行编码与执行算法方面展现出巨大的潜能。此前,研究者利用DNA分子反应网络,成功实现了细胞自动机、逻辑电路、决策机器、神经网络等多种功能。
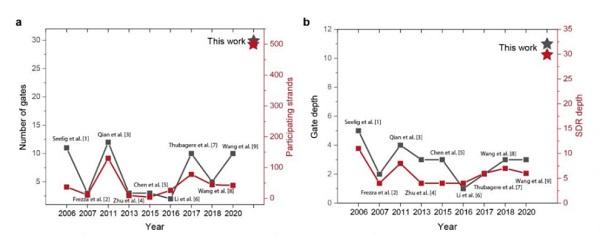
图说:代表近20年来DNA计算领域的新突破
研究团队针对发展具有通用性的DNA运算元件的编程与集成问题,首先证明了利用单链DNA作为统一传输信号(DNA-UTS),可实现类似电子在电路中传输的功能。进而,开发了一种支持通用性数字计算的DPGA,并支持器件层次的多DPGA集成,实现了器件内的可编程性和器件间可集成性。
利用DPGA的可编程性与高集成度,该研究突破了DNA分子计算在电路规模和电路深度的瓶颈,首次在实验上展示了高达30个逻辑元件、500条DNA链,包含30层DNA链取代反应的电路规模。这也代表了近20年来DNA计算领域的新突破。此外,该研究概念性展示了将DPGA作为分子诊断中的信息处理核心,对疾病相关的分子靶标进行非线性分类。
这项工作得到了国家自然科学基金委、科技部、上海市科委、新基石科学基金会等项目的资助。
-
媒体链接