
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大庆睿研究组与合作者共同发布基于隐空间扩散模型的蛋白质序列设计算法PRO-LDM
近日,上海交通大学生命科学技术学院、微生物代谢全国重点实验室庆睿课题组与香港科技大学(广州)许人镜课题组合作在Advanced Science上发表题为PRO-LDM: A Conditional Latent Diffusion Model for Protein Sequence Design and Functional Optimization的研究成果。本工作提出了一种基于隐空间扩散生成模型的蛋白质序列设计方法,通过在局部与全局尺度上学习氨基酸及序列层面的生物学特征,实现类天然蛋白质的多样性设计及理化性质和功能设计,可利用条件引导强度调节在实现隐空间“分布外”采样以实现蛋白质定向优化,并通过设计高亮度、高可溶性和稳定性的绿色荧光蛋白(GFP)变体验证了模型的可用性。生命科学技术学院博士生张司韬与港科技(广州)研究生姜梓轩为共同第一作者,生命科学技术学院长聘教轨副教授庆睿与港科技(广州)助理教授许人镜为共同通讯作者。
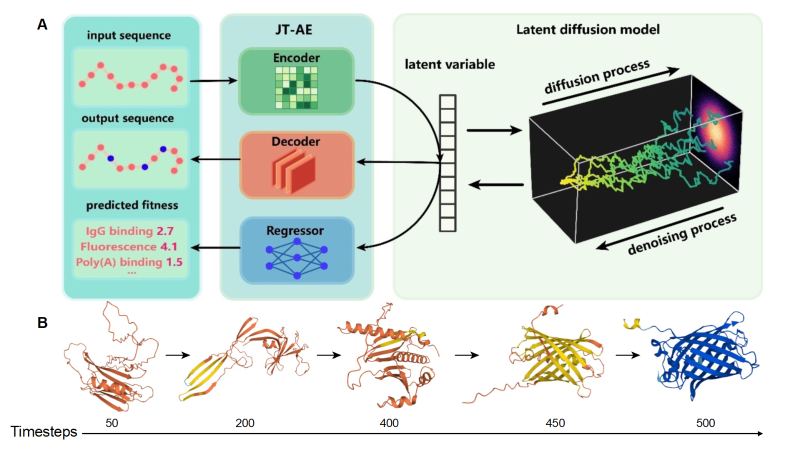
图1. PRO-LDM的模型架构以及扩散生成过程的蛋白结构可视化
蛋白质是维系生命活动的关键分子机器,而天然蛋白仅占据序列空间的一小部分。蛋白质设计旨在主动探索这一空间,通过改造或从头设计序列以拓展功能多样性。计算设计依托日益丰富的蛋白质数据库,较传统理性设计和定向进化更高效准确,降低了对高通量实验的依赖。随着深度学习的发展,蛋白质结构预测与设计迎来了新的范式。由于序列数据量大、信息粒度细,序列设计作为更直接的策略受到广泛关注。本研究提出了名为 PRO-LDM (Protein sequence generation with conditional Latent Diffusion Models)的多任务模块化框架,在隐空间引入扩散模块可兼顾设计精度与效率。该模型支持条件与非条件序列蛋白设计,生成结果多样性高。通过引入适应度标签数据PRO-LDM 可实现目标导向的条件生成与同步预测;在无标签情况下亦可进行无监督生成,产出与天然蛋白功能相似但序列更具多样性的蛋白质。
在条件生成任务中,PRO-LDM 可通过调控引导强度超参数ω实现对生成序列多样性的控制从而生成分布外数据点。当ω调整至 20 时,结合蛋白质适应度预测模型,本研究筛选出一种新型绿色荧光蛋白变体 pro_2421,其荧光强度超过野生型与数据集中表现最优变体;当ω超过 20 时,PRO-LDM 能生成与野生型 GFP 相似度低于 50%、但pLDDT高于 85 的新型突变体,实现全序列高多样性采样。模型采用模块化架构,支持替代集成不同预训练模型以提升扩展性与泛化能力,如在 GFP 设计任务中使用 ESM2编码器可获得更快收敛速度和更优隐变量的特征表示。通过引入结构序列多样性训练集(如CATH或SwissProt),PRO-LDM 可实现不局限于特定蛋白家族的从头设计。相比传统单任务算法,PRO-LDM 在计算效率与设计表现方面更具优势,作为可组合的蛋白质设计工具,能高效提取序列中潜在的生物信息,助力面向结构或功能的蛋白开发,展现出广泛的应用潜力。
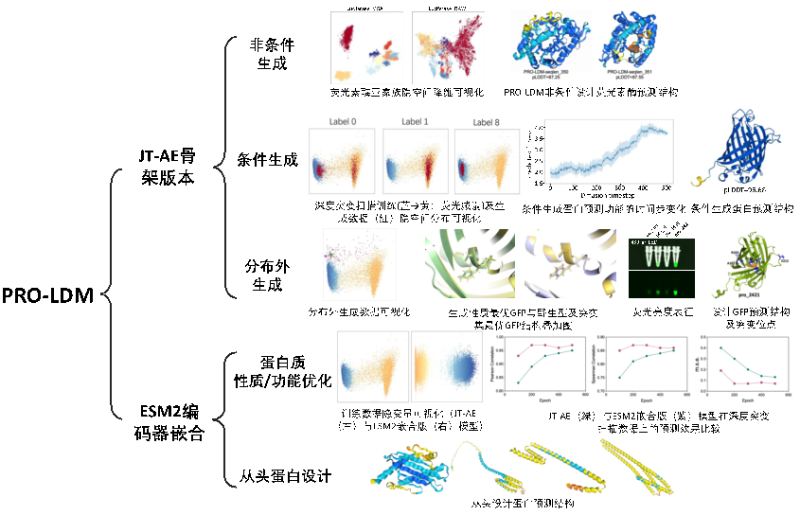
图2. PRO-LDM应用于多种蛋白质设计任务及效果展示
该研究获得国家自然科学基金(32470105)和上海市科学技术委员会(24HC2810800)的资助与支持。同时感谢之江实验室提供的算力和晶泰科技提供的功能筛选模型支持。
庆睿,长聘教轨副教授,微生物代谢全国重点实验室固定研究人员。近年来的研究专注于膜蛋白水溶化设计,及其作为类抗体诱饵受体在医药学以及特异性生物活性探针在生物传感中的应用。目前课题组的主要研究方向包括:(1) 基于深度学习的蛋白设计算法;(2)新型膜蛋白在微生物代谢、医药学和精准治疗领域的应用,和(3)特异性低维分子整合的生物电化学界面等。课题组长期招聘博士后并根据学院指标分配招收硕博研究生,欢迎感兴趣者沟通交流。
论文链接:
https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202502723