
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大曹骎课题组合作揭示细菌生物膜核心蛋白FapC的淀粉样纤维稳定机制及生物材料应用潜力
近日,上海交通大学Bio-X研究院曹骎课题组与丹麦奥胡斯大学Daniel E. Otzen院士团队及广西医科大学第一附属医院王华丙团队合作,在《Advanced Materials》发表了题为“Natural Design of a Stabilized Cross-β Fold: Structure of the FapC from Pseudomonas Sp. UK4 Reveals a Critical Role for Stacking of Imperfect Repeats”的研究成果(图1)。该工作通过冷冻电镜(cryo-EM)首次解析了假单胞菌UK4菌株功能性淀粉样蛋白(Functional Amyloid,FuA)FapC的高分辨率(3.2 Å)结构,揭示了其通过不完全重复单元(Imperfect Repeats, IRs)的异质性堆叠形成稳定希腊钥匙状原纤维的分子机制,为细菌生物膜形成机制及新型生物材料设计提供了结构基础,也是具有IRs的FuA首个能辨别氨基酸残基侧链的高分辨率结构。

图1 发表论文首页截图
FuA是细菌生物膜的关键结构组分,其独特的交叉β折叠构象赋予材料超强稳定性与可编程特性,被视为下一代纳米生物材料的理想模板。然而,由于缺乏高分辨率结构信息,FuA的精确自组装机制及工程化改造长期面临瓶颈。FapC作为假单胞菌生物膜的核心蛋白,由三组IRs(IR1-IR3)构成,但其原子级结构及IRs间的互作模式此前尚未明确。
研究团队通过冷冻电镜三维重构技术,成功解析了FapC原纤维的精细结构,发现其单体通过希腊钥匙状折叠形成三层交叉β结构,每层对应一个IR单元(图2)。结构分析表明,保守的Asn和Gln残基通过密集氢键网络维持希腊钥匙构象,而IRs间的疏水核心残基共进化则确保了IRs堆叠的协同性。有趣的是,IRs的异质性堆叠不仅降低了电荷排斥,还赋予原纤维独特的应变依赖性纳米力学特性。进一步的突变实验证实,破坏关键氢键网络会显著增加纤维断裂倾向,但最终纤维仍保持与野生型相当的稳定性,揭示了FapC结构设计的容错性。
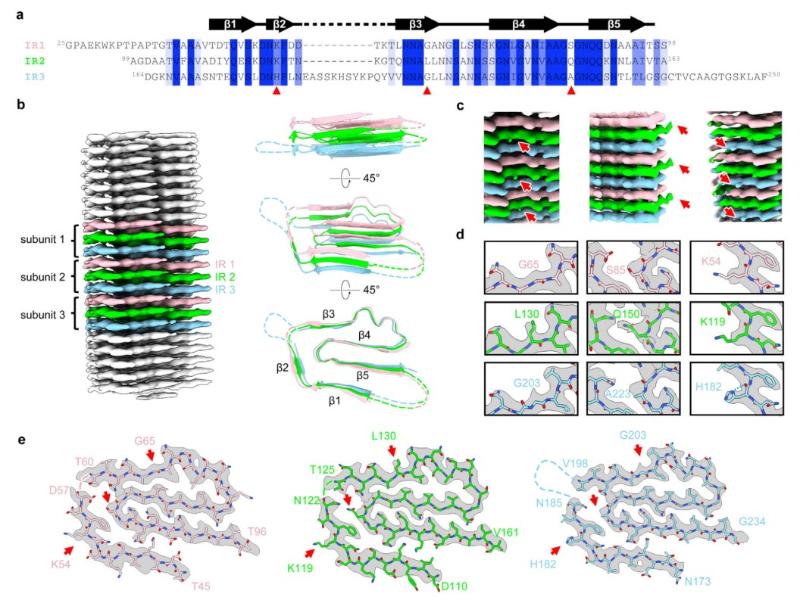
图2 论文中报道的FapC纤维结构
更引人注目的是,FapC纤维展现出本征酯键水解催化活性,且在不同极端条件(如高温、强酸碱)下仍保持活性,凸显了其作为功能性生物材料的潜力。研究还比较了不同假单胞菌株FapC的机械性能,发现UK4菌株的“极简”结构(仅含核心淀粉样区域)具有更高的刚性,而含长链连接区的菌株纤维更柔软且宽度更大,这为材料性能的定向调控提供了新思路。
此项工作首次在原子层面阐明了细菌FuA的自组装密码,不仅揭示了IRs的协同折叠机制,还验证了淀粉样纤维作为催化平台的可行性。研究提出的“异质性堆叠稳定化”模型为人工设计高稳定性生物材料提供了全新策略,未来或可应用于环境修复、纳米催化及生物医学工程等领域。
广西医科大学与上海交通大学联合培养博士研究生蒋艳婷为本论文共同第一作者,另两位共同第一作者分别为丹麦奥胡斯大学博士后Samuel Peña-Díaz和广西医科大学博士后张哲菲 。上海交通大学Bio-X研究院曹骎副教授为本论文共同通讯作者。
论文链接:https://doi.org/10.1002/adma.202505503