
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大张宏陆团队在发展框架核酸增强的单分子光镊技术方面取得进展
近日,上海交通大学自动化与感知学院张宏陆副教授团队与中国科学院院士、上海交通大学樊春海教授,美国科学院院士、加州大学伯克利分校Carlos Bustamante教授合作,在《Science Advances》期刊上发表了题为 “DNA Origami-enhanced Force Spectroscopy and AlphaFold Structural Analyses Reveal the Folding Landscape of Calcium-Binding Proteins”(框架核酸增强的单分子力谱技术与AlphaFold结构解析揭示钙结合蛋白折叠动态图景)的最新研究成果。该工作利用框架核酸增强的单分子光镊技术,结合 AlphaFold 结构分析,揭示了钙结合蛋白(CaE)紧凑多结构域蛋白的复杂折叠路径及“隐藏”中间态,为理解蛋白质折叠机制提供了新视角。

研究背景
分析和阐明蛋白质如何正确折叠,对于理解其动态机制和相关疾病发生发展、开发疾病诊断和治疗方法至关重要。近年来,AlphaFold等深度学习方法在预测蛋白质静态结构方面取得了突破性进展,展示了人工智能在生命科学领域(AI for Science)的巨大潜力。然而,现有的AI技术仍难以对蛋白质折叠的动态过程进行准确预测。
创新成果
研究团队利用DNA折纸术构建了具有六方密堆积排列的框架核酸结构,其力学刚性较传统双链DNA提升8倍,可取代后者作为高精度单分子力学传感器,其应用显著提高了光镊测量的时空分辨率,成功捕捉到了以往研究中被忽略的钙结合蛋白折叠中间态。实验结果表明,框架核酸结构具有很高的机械刚性,可有效降低测量噪声。特别是在低力区域(<5 pN),测量信噪比可提升近一倍,这使得研究人员能够实时观测蛋白质折叠和展开的全轨迹过程,包括瞬时随机出现的中间态。
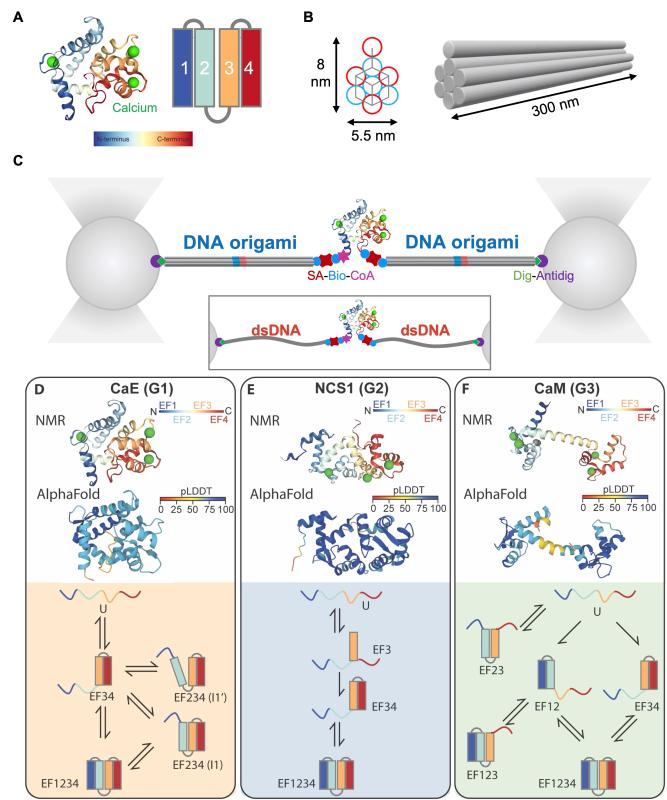
框架核酸增强光镊+AlphaFold揭示蛋白折叠动态图景:(A)钙结合蛋白CaE(B)框架核酸刚性力学传感器(C)单分子光镊操控装置 (D-F)钙结合蛋白家族三类蛋白(CaE、NCS1和CaM)的结构预测和动态折叠路径
通过与AlphaFold结构预测相结合,研究团队对钙结合蛋白家族的折叠行为进行了系统比较分析。研究发现,尽管钙结合蛋白家族成员虽具有相似的EF-hand结构基序,但因连接区域长度和柔性的差异,展现出独特的折叠路径和动力学特征。此外,该研究还揭示了钙结合蛋白内部的疏水相互作用、氢键网络等非共价相互作用在维持其稳定性和折叠路径中发挥着关键作用。
该研究融合框架核酸单分子技术与深度学习驱动的结构预测方法,展现了“AI for Science”理念中实验技术与计算预测的协同优势,这一创新方法为解决复杂生命科学问题提供了新的研究范式,有望进一步推动人工蛋白质设计与合成生物学领域的研究。
文章信息
张宏陆与美国斯坦福大学Cristhian Cañari-Chumpitaz博士为该论文的共同第一作者;张宏陆、樊春海与Carlos Bustamante为论文的共同通讯作者。该研究工作得到了国家重点研发计划、国家自然科学基金等项目的资助。
论文链接:https://www.science.org/doi/epdf/10.1126/sciadv.adv1962
研究团队介绍

张宏陆研究团队长期致力于单分子生命感知、人工智能与合成生物学、DNA信息存储等前沿领域的研究,相关研究成果以第一/通讯作者发表于《Science Advances》《Nature Communications》《PNAS》《Matter》《Nature Protocols》等高水平学术期刊,申请发明专利十余项,并积极开展颠覆性技术的转化研究。欢迎感兴趣的同仁加入团队共谋发展!
联系邮箱: z.hl@sjtu.edu.cn