
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大毛亚飞课题组与合作者Nature发文解析猕猴属复杂结构变异助力演化医学研究
猕猴属的食蟹猴(Macaca fascicularis)和恒河猴(Macaca mulatta)是最常用的非人灵长类实验动物,因其与人类具有较近的亲缘关系(约2500万年前分化),而被广泛用于生物医学和人类演化研究,尤其在人类特有性状形成、疾病模型及药物代谢等领域。然而,现有的参考基因组仍存在大量未知序列,特别是着丝粒、片段重复(segmental duplications, SDs)和核糖体 DNA(rDNA)区域,这限制了对灵长类演化机制及其生物医学价值的深入探索。

图 1 Nature论文首页截图
2025年2月26日,上海交通大学Bio-X研究院毛亚飞课题组与中国科学院脑科学与智能技术卓越创新中心/神经科学研究所孙强课题组合作,在Nature期刊上发表了题为Integrated analysis of the complete sequence of a macaque genome的文章(图1)。该研究首次完成了非人灵长类端粒到端粒(telomere-to-telomere, T2T)完整基因组组装,系统解析了猕猴属与人类的大尺度基因组差异,并以FOLH1基因家族为关键案例,解释结构变异如何通过三维基因组重构调控脑细胞类型特异性表达。除此之外,该项研究还揭示了猕猴属种间分化的遗传学特征,为非人灵长类生物医学模型奠定了关键遗传基础。
1. 填补复杂区域空白
传统基因组组装技术因读长短、错误率高,难以跨越复杂重复区域,导致基因组中存在大量缺口。这些缺口多位于基因组复杂结构区域(着丝粒、SD 和 rDNA 等),而这些区域恰恰是基因组结构和功能的重要部分,涉及染色体的稳定性、基因表达调控以及染色体的重排等生物学过程。
为了解决这一难题,研究团队首先构建了食蟹猴孤雌生殖胚胎干细胞系(MFA582-1)【1】。孤雌生殖细胞系具有两套染色体遗传信息近乎纯合的特性,是构建参考基因组的理想材料。在此基础上,团队利用自主开发的基于特有k-mer标记的分型迭代替换局部组装工具,成功解决了现有组装软件未能或错误组装的上百个复杂结构区域,最终构建食蟹猴T2T基因组T2T-MFA8v1.1。该基因组全长3.06Gbp,QV达到71.27,NG50为162.13Mbp,与人类基因组T2T-CHM13v2.0的水平相当,成为首个非人灵长类完整参考基因组,为深入理解复杂基因组区域提供了重要工具。研究团队发现:
SD分布模式的特异性:食蟹猴的SDs总长122.51Mbp,较人类(227.36Mbp)减少46%,且更富集于亚端粒区(人类偏向近着丝粒区)。这种分布差异可能驱动灵长类基因组重塑的方向性。
rDNA分布:食蟹猴的rDNA仅定位于10号染色体,而在人类基因组中,rDNA分布于5条近端着丝粒染色体(chr13, chr14,chr15, chr21, chr22)。这一差异为人类近端着丝粒染色体的形成及人类疾病21三体综合征的演化医学机制提供了新的解释。
着丝粒结构演化:食蟹猴着丝粒由SF7家族α-卫星主导,平均长度较人类长3.83倍(如chr15达13.88Mbp),并保留与灵长类祖先共存的古老SF8-SF13序列层,揭示了猕猴属存在新着丝粒形成的独特演化机制。
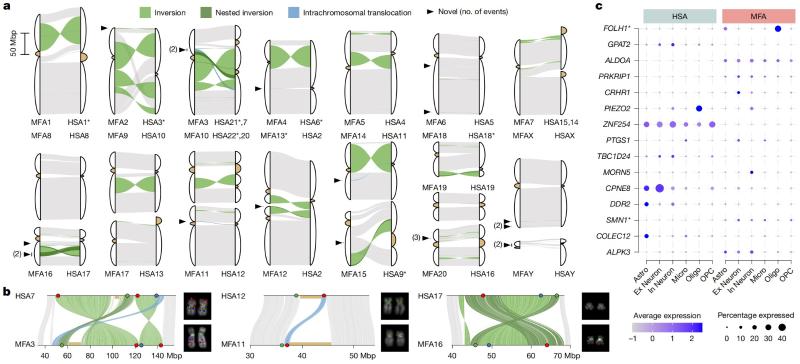
图 2 人类和猕猴属的固定大尺度结构变异
2.人类与猕猴属大尺度结构变异导致的基因组差异
基因组的大尺度结构变异(如倒位、易位)能够改变染色质三维折叠,进而影响基因表达模式。然而,人类与猕猴属之间的大尺度基因组结构变异尚未得到全面解析,且这些结构变异在灵长类演化过程中的功能效应仍缺乏系统性的证据支持。
为解决这一问题,研究团队优化了结构变异鉴定方法,共鉴定出93个人类与猕猴属之间的固定结构变异,其中包括78个倒位、11个着丝粒重定位和4个染色体内易位,其中21个结构变异为首次报道(图 2)。进一步研究发现,在灵长类脑演化过程中,超过400个基因可能因相关结构变异而发生脑细胞类群的表达差异。以FOLH1基因为例,其编码的谷氨酸羧肽酶II(glutamate carboxypeptidase II,GCPII)在神经系统的谷氨酸调控中发挥关键作用,基因突变与智力障碍密切相关【2】。在猕猴属中,FOLH1基因为单拷贝,而在人类中,则包含FOLH1和 FOLH1B(与猕猴FOLH1直系同源)两个拷贝。人类的FOLH1基因在少突胶质细胞中高表达,而FOLH1B基因在大脑不同细胞类群中几乎不表达,与猕猴属FOLH1在多个细胞类群中广泛表达的模式存在显著差异。为了揭示这种差异的成因,研究团队整合了单细胞多组学数据,发现在人类与猕猴属的演化过程中,人类FOLH1B调控区的一个1.4 kbp片段特异性地丢失,导致了其在脑内无法表达而“假基因化”。同时,人类的FOLH1基因由于重复事件的发生,导致了染色质三维结构的重塑,进而改变了其细胞类群表达模式(图3)。这一研究为结构变异在演化过程中如何影响细胞类型特异性表达模式提供了新的见解,特别是在阐明谱系特异性表型的形成及人类疾病的发生机制解析上具有重要的科学意义。
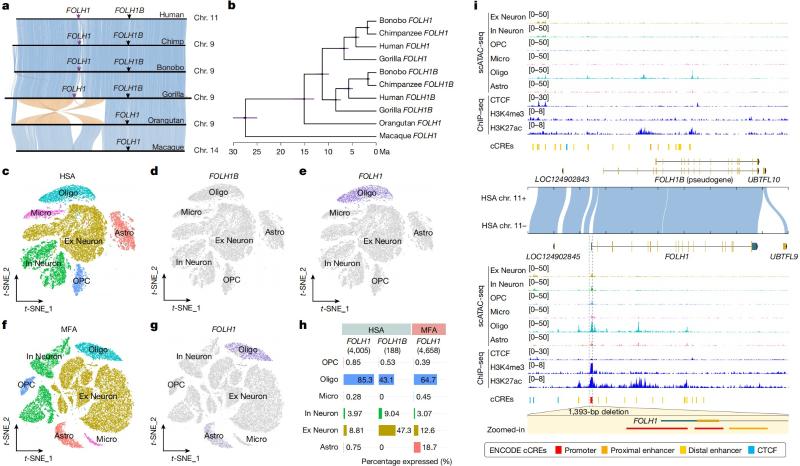
图 3 FOLH1 基因家族的演化历史和多组学分析
3.食蟹猴与恒河猴间从序列到表型的种间遗传分化
食蟹猴与恒河猴虽同属于猕猴属,但在形态学特征、行为模式及疾病易感性等方面均存在显著差异。然而,这些表型差异背后的遗传基础一直未得到充分阐明,从而严重制约了这两个物种在生物医学模型中的应用潜力。
研究团队通过泛基因组图等手段,鉴定了240Mbp的物种间复杂结构差异区域,涵盖Mafa-AG/B、CYP2C76及GSTM等基因家族。其中,恒河猴CYP2C76(猴特异性细胞色素P450酶基因)呈现4种结构单倍型,而食蟹猴仅保留2种,这可能反映了两个物种之间的代谢差异(图4)。此外,研究团队鉴定出16.76Mbp的食蟹猴与恒河猴遗传分化区域(genetic differentiation regions)(图4),发现两物种间的固定遗传差异在调控元件中出现了9.43倍富集,其中包括可能影响尾巴长度的HOXD13基因。这一发现与食蟹猴尾巴显著长于恒河猴的表型特征相吻合。此外,该发现也与鹿鼠尾巴适应性演化的研究【3】形成了趋同演化案例,为揭示哺乳动物尾巴形态多样性的遗传机制提供了新的视角。
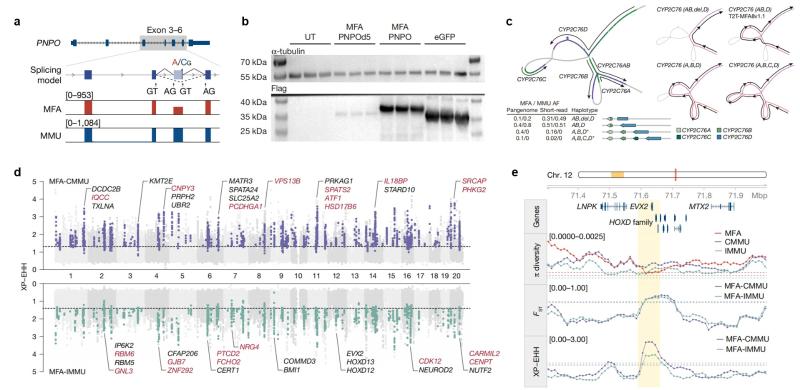
图 4 食蟹猴与恒河猴的种间遗传分化
总体而言,本研究通过研发新型计算工具实现非人灵长类T2T完整基因组组装,系统阐明了猕猴属与人类在基因组结构层面的演化差异,不仅揭示了结构变异通过三维基因组重构调控和调控元件改变等影响基因表达的细胞类型特异性,还深入解析了猕猴属种间分化的遗传学基础,为猕猴生物医学模型奠定了坚实的遗传基础。该研究深化了我们对灵长类演化医学、生物医学模型和谱系特异性适应的理解。
该工作是在上海交通大学Bio-X研究院支持下的毛亚飞团队一系列研究成果中的代表性工作。上海交通大学Bio-X研究院贺林院士指出,“本项成果是染色体大规模变异研究的一个经典案例,相关工作为解析灵长类基因组复杂结构变异的生物医学功能提供了全新视角,推动了信息学、演化生物学与医学遗传学的交叉发展,为演化医学领域的未来研究奠定了重要基础”。
上海交通大学Bio-X研究院毛亚飞与中国科学院脑智卓越创新中心孙强为该论文的共同通讯作者。该工作得到了上海交通大学、中国科学院脑智卓越创新中心、浙江大学、中国科学院昆明动物研究所、中国科学院成都生物研究所等专家学者的鼎力支持。上海交通大学和中国科学院脑智卓越创新中心张世龙、许宁和傅莲婷为该论文的共同第一作者。本研究与国内外多个灵长类联盟合作,在此感谢联盟内的数据共享支持。
上海交通大学Bio-X研究院毛亚飞研究组专注灵长类演化医学研究,以演化生物学、计算生物学、神经生物学和大规模功能筛选等交叉学科手段解析灵长类特有适应性性状形成和人类疾病风险位点发生的遗传机制。研究组长期招收/聘具有演化生物学、生物信息学、细胞生物学、神经科学、计算科学等多学科背景的研究生、博士后和研究助理,为有志于从事灵长类演化医学研究的优秀人才提供广阔的发展平台。如果您对该研究组的研究方向感兴趣,并具备相关专业背景和技能,欢迎访问研究组介绍网站(https://www.yafmao.org/)了解更多信息。
论文链接:https://www.nature.com/articles/s41586-025-08596-w
参考文献:
1. Yang, H. et al. Generation of haploid embryonic stem cells from Macaca fascicularis monkey parthenotes. Cell Research 23 (2013). https://doi.org/10.1038/cr.2013.93
2. Rahn, K. A. et al. Inhibition of Glutamate Carboxypeptidase II (GCPII) activity as a treatment for cognitive impairment in multiple sclerosis. Proceedings of the National Academy of Sciences 109 (2012). https://doi.org/10.1073/pnas.1209934109
3. Kingsley, E. P. et al. Adaptive tail-length evolution in deer mice is associated with differential Hoxd13 expression in early development. Nature Ecology & Evolution (2024).
https://doi.org/10.1038/s41559-024-02346-3