
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大钱晓华课题组基于因果创新开创甲基化生物标志物挖掘新突破
DNA甲基化生物标志物挖掘近年来在疾病早期诊断、分子机制研究和风险预测等领域展现出巨大潜力。然而,定义潜在的因果关系是一项重大挑战,尤其混杂因素(如测量噪声和个体差异)的干扰导致现有方法生成的候选标志物可靠性不足,进而需要依赖昂贵且耗时的实验验证。这不仅限制了研究的广泛开展,也阻碍了临床应用的推进。
为此,上海交通大学生物医学工程学院钱晓华课题组近日在《Nature Communications》期刊上发表文章Causality-driven Deep Regularization for Reliable DNA Methylation Biomarker Discovery,从因果关系角度排除噪声干扰、识别潜在致病因素,为DNA甲基化生物标志物的可靠挖掘提供了全新的解决方案,有效解决了传统方法在因果关系推断中的局限性,显著提升了生物标志物发现的准确性和可靠性。唐欣鹭、郭睿和莫湛锋为该论文的共同第一作者,钱晓华老师是唯一通讯作者。

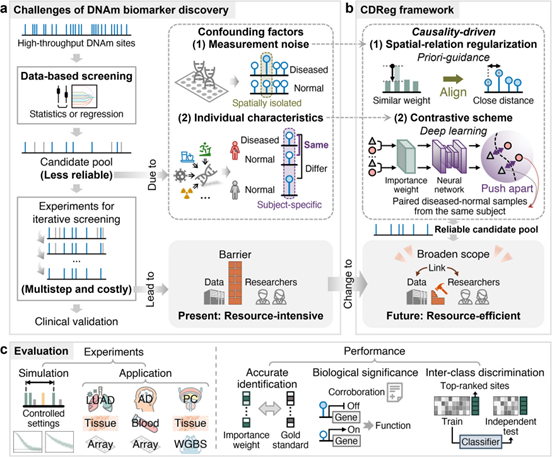
图1 本研究的概述
a:DNA甲基化生物标志物挖掘的挑战;b:所提出的因果驱动的深度正则化(CDReg)框架概述;c:综合性能评估。
作者开发了一种因果驱动的深度正则化框架,整合因果思维、深度学习和生物学先验知识,通过两项关键创新解决甲基化标志物挖掘可靠性不足的难题。具体来说,1)提出空间关系正则化:利用共甲基化特性,鼓励相邻位点获得相似权重,从而排除孤立噪声位点,优先选择聚集性位点簇。2)开发对比方案:受随机对照试验启发,将同一主体的患病与正常样本在嵌入空间中分开,放大疾病特异性位点的权重,确保所选标志物真正与疾病相关。
实验结果表明,所提出的框架在模拟数据和大型公开数据(如肺腺癌、阿尔茨海默病和前列腺癌)中均表现出色,能够有效识别具有强类间鉴别性和生物学相关性的生物标志物。此外,该框架可灵活处理多种数据类型(如微阵列和全基因组亚硫酸氢盐测序数据),为研究人员提供了强大的工具支持。
研究团队与本院古宏晨-徐宏教授团队长期密切合作,专注于DNA甲基化癌症早筛技术的研发,负责其中数据挖掘算法设计,打通了从生物标志物发现1,2到引物设计3和检测技术4的全面流程。最具影响力的成果之一是结直肠癌早期筛查工具,已成功应用于万人级别的大型前瞻性队列研究,并在合肥社区筛查中成功检测出多例早期癌症和癌前病变,为及时干预和治疗提供了宝贵的时间窗口。通过结合生物信息学、分子生物学和临床专长的跨学科合作,团队成功将计算发现转化为实际应用,助力公共健康事业发展。未来,团队计划将成果扩展至更多疾病领域,推动早期检测和精准医疗的普及,赋能健康中国建设。
研究组介绍
上海交通大学生物医学工程学院钱晓华课题组(Medical Image and Health Informatics Lab,MIHI,https://mihi.sjtu.edu.cn)长期致力于重大疾病微小进展的前沿研究,围绕肿瘤和运动障碍开发一系列微小特征挖掘与稳定分析算法,并将科研成果转化为实际应用。除上述通过革新数据挖掘策略实现癌症甲基化高效早筛之外,主要成果还包括:1)提出医学动作视频细分技术,开创利用动作视频实现帕金森病智能评估的新体系;2)提出低对比度肿瘤影像泛化性表征技术,首创体检级CT实现胰腺癌筛查与早诊的基层可行新模式;3)提出复杂微小器官的影像稳定性分析技术,提供利用临床级影像实现胰腺癌精准手术规划的新范式。基于上述共性通用技术,成功构建了帕金森病视频智能评估系统和胰腺癌“筛-诊-疗”全栈式智能生态,服务于广大患者。
参考文献
[1] Xinlu Tang#, Rui Guo#, Zhanfeng Mo#, Wenli Fu, Xiaohua Qian*. Causality-driven candidate identification for reliable DNA methylation biomarker discovery. Nature Communications, 2025, 16: 680.
[2] Xinlu Tang#, Zhanfeng Mo#, Cheng Chang, Xiaohua Qian*. Group-shrinkage feature selection with a spatial network for mining DNA methylation data. Computers in Biology and Medicine, 2023, 154: 106573.
[3] Gaolian Xu#, Hao Yang#, Jiani Qiu#, Julien Reboud, Linqing Zhen, Wei Ren, Hong Xu*, Jonathan M. Cooper*, Hongchen Gu*. Sequence terminus dependent PCR for site-specific mutation and modification detection. Nature Communications, 2023, 14: 1169.
[4] Linqing Zhen#, Xinlu Tang#, Zhengguo Xu#, Yizhou Huang, Xiaohua Qian, Haiping Lin, Chao Li, Rong Cui, Hongsheng Fang, Hao Yang, Jiani Qiu, Zhaoqi Fang, Xiaohuan Peng, Yifeng Jin, Jianing Nie, Shiwei Guo, Yuguang Wang, Ming Zhong*, Hongchen Gu*, Hong Xu*. Early Diagnosis of Colorectal Cancer Based on Bisulfite-free Site-specific Methylation Identification PCR Strategy: High‐Sensitivity, Accuracy, and Primary Medical Accessibility. Advanced Science, 2024: 2401137.
论文链接:https://www.nature.com/articles/s41467-025-56054-y