
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大宋萍课题组在JACS发表智能核酸探针选择性擦除方法用于DNA数据存储
近日,上海交通大学生物医学工程学院宋萍团队在Journal of the American Chemical Society期刊上发表题为“Random Sanitization in DNA information storage using CRISPR-Cas12a”的最新研究。该研究开发了一种智能核酸探针杂交精准调控与CRISPR-Cas12a反式切割活性的选择性永久擦除方法(RSDISC),可用于实现存储信息的加密与保护,并在包括三字经、道德经、孙子兵法、I have a dream、上海交通大学庙门图片等多模态DNA存储近三万条序列上得到验证。
随着互联网、人工智能及其他信息技术的飞速发展,全球数据量预计将在2025年达到175 ZB。如何实现高效的大数据存储已成为亟待解决的瓶颈问题。DNA数据存储作为一种新兴的存储技术,凭借其超高的存储密度、安全性及长期稳定性,被广泛认为是应对大数据存储需求的理想方案。然而,尽管DNA存储展现出巨大潜力,当前在数据安全方面仍面临诸多挑战。特别是在大数据存储中,如何设计高度安全的加密系统以保护敏感信息,及如何实现数据的永久删除,依然是亟待攻克的难题。
上海交通大学宋萍课题组构建了一种基于Cas12a反式切割活性和引物-模板智能核酸选择性杂交的方法实现了在DNA存储中高灵敏、高特异性地永久擦除目标信息(图1)。该方法通过智能核酸杂交设计及调控,构建目标文件对应的反向引物选择性杂交并扩增为双链,从而实现对目标文件的保护;同时,激活的Cas12a复合物切割未被保护的单链DNA,实现数据的精确擦除。通过进一步优化核酸杂交热力学和Cas12a切割活性的条件,研究者在由28,258条寡核苷酸组成的包括图片和文字在内的多模态DNA存储体系中验证了该方法具有高达99.9%的擦除效率和99.5%的特异性。该方法不仅可用于保护敏感数据,还能在大数据存储过程中实现内存清理、文件分类,并提高测序准确性,并可拓展在分子诊断等领域的广泛应用。
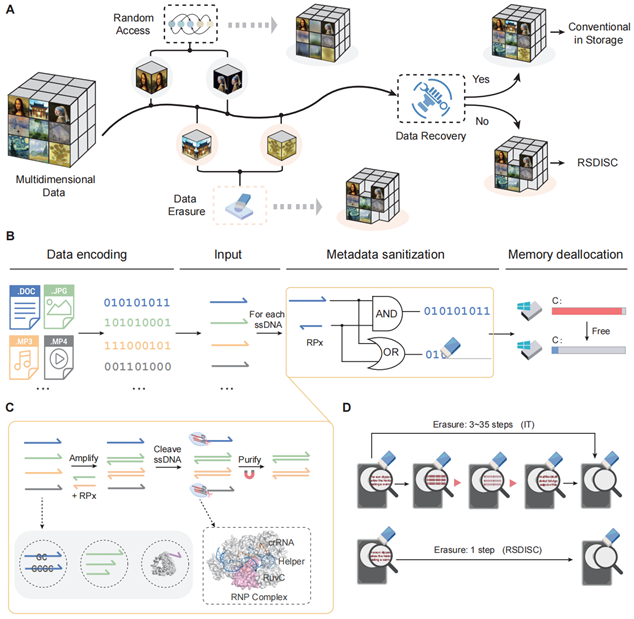
图1 选择性擦除方法(RSDISC)的工作流程图
研究者首先在单重体系中验证了该方法的可行性(图2)。通过设计两种Cas12a激活体系以评估其对单一模板的切割效率,并进一步分析了这两种体系在不同GC含量、长度和复杂二级结构修饰的单链DNA上的表现。实验结果表明,该方法能够高效切割多种类型的单链DNA,切割效率最高可达99%。随后,通过将该方法应用于具有模板相互作用的复杂多重体系,发现所有模板的切割效率均超过90%(图3)。此外,为评估RSDISC技术在大规模数据清理中的应用潜力,基于热力学杂交原理的模拟结果表明该方法能擦除高达158亿个文件,相当于10 PB的数据量。
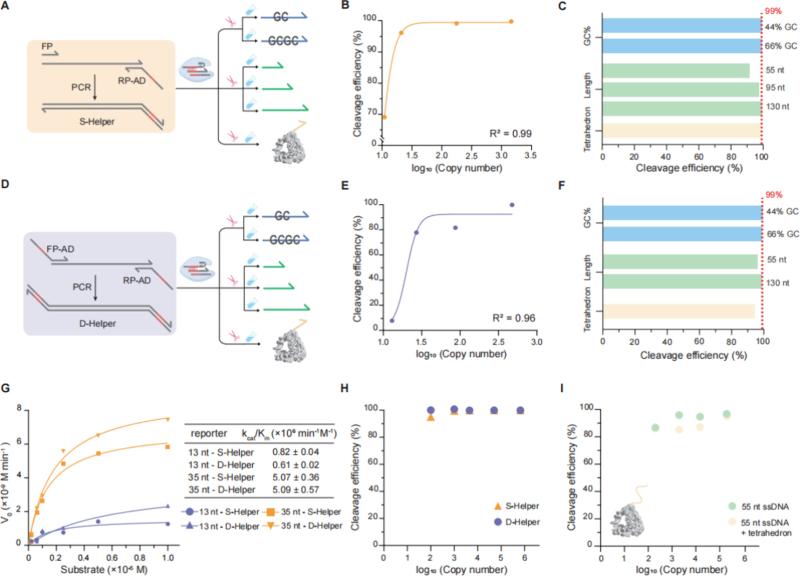
图2 单重体系中RSDISC方法的切割效果
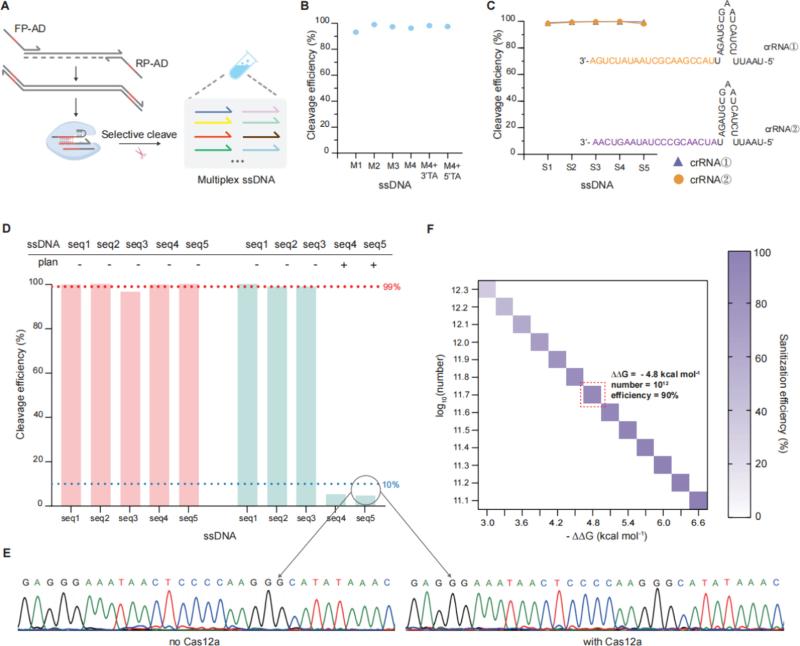
图3 多重体系中RSDISC方法选择性切割的效果
为了验证RSDISC方法在实际存储体系中的效果,作者进一步编码并存储包括三字经、孙子兵法、道德经、上海交通大学庙门图片、蒙娜丽莎等在内的七个文件于近三万条DNA序列中。实验结果表明,RSDISC方法的寡核苷酸擦除效率高达99.9%,擦除特异性为99.5%(图4),有效证明了该方法在高效擦除非目标文件信息的同时,几乎不会影响靶向保留的信息。这项工作为DNA存储提供了高效可靠的信息加密方案,未来将在大规模数据存储和提升数据处理效率方面发挥重要作用。
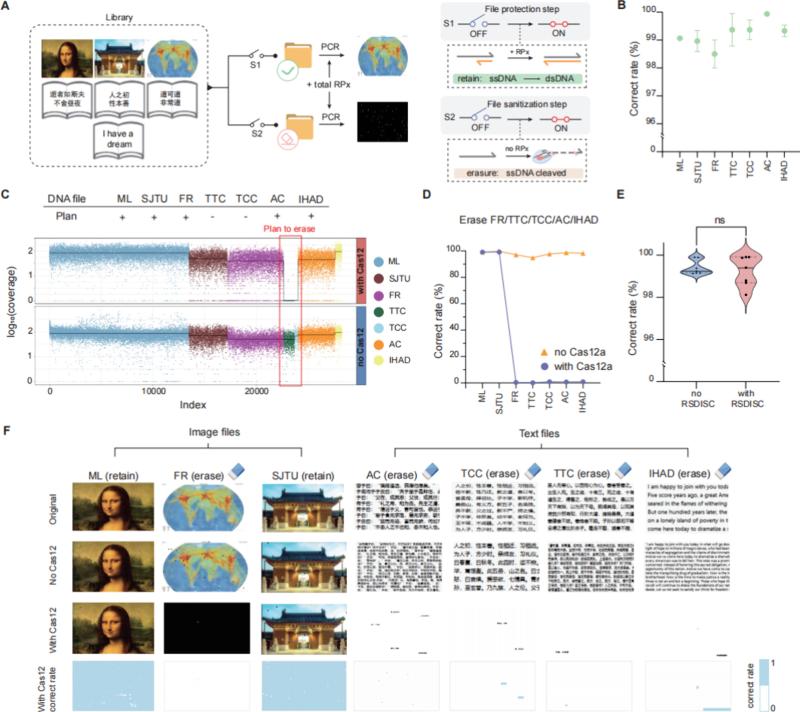
图4 实际存储体系中选择性擦除文件的效果
上海交通大学生物医学工程学院博士研究生沈虹雨是该论文的第一作者,宋萍副教授为通讯作者。本工作得到了国家重点研发计划、国家自然科学基金、中央高校基本科研业务费、上海市教育委员会“青年领军人才培养计划”项目等的资助。
论文连接:https://pubs.acs.org/doi/10.1021/jacs.4c11380