
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大洪亮课题组发表基于微环境感知图神经网络构建指导蛋白质定向进化的通用人工智能的最新研究成果
近日,上海交通大学自然科学研究院/物理与天文学院/张江高等研究院洪亮课题组设计了一种微环境感知图神经网络(ProtLGN),能够从蛋白质三维结构中学习有益的氨基酸突变位点,建立自然选择下的氨基酸序列分布,用于指导蛋白质氨基酸位点设计,最终实现蛋白质指定功能的提升(定向进化)。课题组通过生物化学室实验证明,这是一个通用人工智能技术,依据目标蛋白极少实验数据甚至没有实验数据的情况下成功实现了多款蛋白指定性质的定向进化(包括抗体的亲和力/稳定性,多个荧光蛋白的荧光强度、核酸内切酶的DNA切割活性)。此外,ProtLGN能够根据单位点突变体活性准确预测多位点组合突变体活性,并在单轮湿实验中即可筛选出活性显著优于低位点突变体的高位点突变体,表明ProtLGN能够有效挖掘蛋白质定向进化中的正上位效应。因此,相较于传统的蛋白质设计方法,ProtLGN能够极大地提升蛋白质设计效率并降低实验成本。该研究成果以“Protein Engineering with Lightweight Graph Denoising Neural Networks”发表于美国化学学会出版社下Journal of Chemical Information and Modeling。
人工智能的快速发展改变着生命科学领域的研究方法和思维范式。其中,蛋白质设计作为生物医药领域的关键技术之一,受到了学界和工业界的广泛关注。传统的蛋白质设计方法通常依赖于试错(蛋白质定向进化)和经验积累(蛋白质理性设计),这种方式虽然在一定程度上能够取得成功,但也存在着效率低下、成本高昂、时间耗费长等难以解决的问题。随着深度学习的快速发展和广泛应用,为蛋白质设计领域注入了新的活力和希望。深度学习模型可以通过大量的生物信息学数据进行训练,并从中学习蛋白质序列、结构与功能之间的关联,为蛋白质设计提供准确、高效的指导。
近几年,基于深度学习的预测与筛选方法被提出并在实际应用中得到了验证与应用。然而,当前的方法大多是基于多序列比对(MSA)和/或蛋白质语言模型(PLM)对蛋白质序列进行特征提取。前者提取出的蛋白质共进化信息的质量高度依赖同源信息的数量,但在实际应用中,并非所有蛋白质序列都能进行同源比对,也并非所有比对都足够深以训练足够大的模型以学习氨基酸之间的复杂相互作用。后者来源于自然语言处理,因此模型主体通常需要搭建Transformer,递归神经网络,或其他自回归模型,这类方法通常需要海量的训练数据和大型且复杂的模型设计,从而需要非常高的训练成本。即使是使用当前主流的语言预训练模型的思路,考虑到每个蛋白质都有独特的性质和进化方向,不涉及任何的重新训练路径而直接把一个预训练好的模型推广到任意的任务中,对于大模型的泛化性和表达能力也是一个巨大的挑战。
因此,本文设计了一款能够提取氨基酸周围微观环境信息的等变图神经网络的预训练框架ProtLGN,结合蛋白质的结构信息对一条蛋白质上的每个氨基酸进行同步编码,学习蛋白质三维结构中有益的氨基酸突变位点和突变类型,用于指导具有不同功能的蛋白质单位点突变和多位点突变设计。
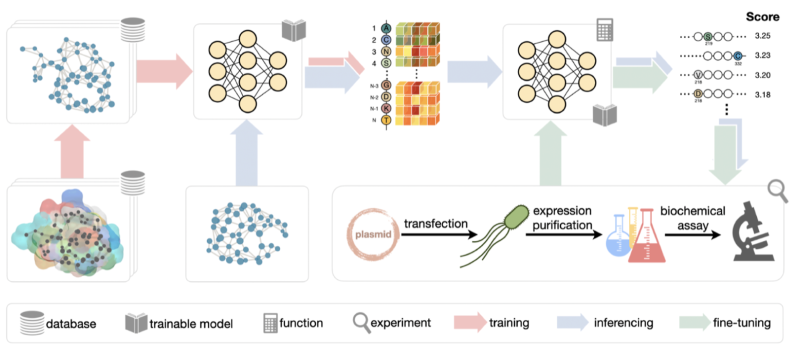
图1 ProtLGN框架示意图
首先,由于蛋白质的序列决定了结构,结构又决定了功能,作者在输入数据时充分使用了蛋白质的结构信息。利用氨基酸序列的三维结构、每个氨基酸的物理和生化性质,以及它与周围不同尺度下的邻居氨基酸的相互关系,作者创立了蛋白质的图表示。在一个蛋白质图上,每个节点表示一个氨基酸,每个氨基酸根据k临近矩阵算法,与它在欧氏距离上最近的最多10个节点相连。作者还综合考虑了单一氨基酸的性质,比如它的氨基酸类型,溶剂可及面积(SASA),B-factor等、前后相邻氨基酸的几何关系(比如二面角,本地坐标系等)、周围1阶邻居氨基酸的相互作用力,以及邻近氨基酸在蛋白质序列上的相对位置等,并利用这些不同尺度上的微观信息来定义图节点和边上的特征。
对于蛋白质的表征学习,作者使用具有旋转和平移等变性的图神经网络。根据物理学定律,无论蛋白质如何从一个地方平移或旋转到另一个地方,原子受到的力都应该保持不变。因此,为了尊重氨基酸的空间关系,也就是旋转和平移等变性,应将对称性的归纳偏差纳入基于蛋白质结构的模型设计中。一种直接的方式是类似图片处理类任务中常用的预处理方法,对输入数据进行增强。对于结构数据来说,另一种方式是针对性地设计等变神经网络,把蛋白质图的节点特征、连接方式,以及三维坐标都输入进模型,通过一系列具有等变性的信息传导网络层,对数据特征进行提取与更新。
另外,为了进一步利用生物学的先验信息来提高模型的泛化性和表达能力,作者还采取了三个额外的措施,包括:对输入的氨基酸类型进行加噪来模仿自然界中的随机突变;在氨基酸节点预测的损失函数打分机制中引入标签平滑来鼓励同类氨基酸之间的置换;利用多任务学习策略让预训练模型学习多种预测目标从而训练一个“一词多用”的图表示学习模型。
LGN的零样本学习训练框架如上图所示。首先,输入蛋白质数据集中的每个序列被k临近邻居算法转换成一个蛋白质图,并基于氨基酸性质提取出节点特征、边特征,以及氨基酸的三维坐标信息。接着,对一部分的节点特征进行噪声扰动后输入到等变图神经网络中学习图上的节点表示。这一节点表示被全连接层解码后可以预测多个不同的目标,比如去噪的节点氨基酸类型标签,SASA和B-factor数值等。这里的预测误差用于构建损失函数并传导回网络层进行反向传导。
在预测阶段,为了得到一个突变体的突变性质分数,作者首先把突变蛋白质的结构输入到预训练好的模型中得到每个节点的氨基酸类型概率。接着,作者把突变体和野生型蛋白质进行比对,并提取出不同部分的氨基酸,以及氨基酸类型对应的联合预测概率。这些数据进行对数处理、变换、加和后,结果的分数就是这个突变体的最终评分。
为了验证ProtLGN对蛋白质突变体活性的预测效果,本文在不同蛋白质的多种生理功能性质上进行了充分验证,确保ProtLGN预测效果的通用性,包括VHH抗体、荧光蛋白(绿色、蓝色和橙色)、核酸内切酶(KmAgo)等多种蛋白的热稳定性、结合能力、荧光强度、单链DNA剪切活性等蛋白质工程常规改造的多种功能。湿实验结果表明,ProtLGN可以在没有湿实验数据或仅少量类似功能蛋白质的实验数据基础上达到40%的单点位改造成功率,并且在部分单位点上实现了多种功能协同提升。上述结果表明ProtLGN能够极大改善传统蛋白质工程方法中成本高、成功率低、数据稀缺等问题。更为重要的是,本文首次使用深度学习模型在学习单位点突变体活性数据后,准确预测组合位点的活性,并且在单轮湿实验中即可筛选出功能显著优于低位点突变体的高位点突变体,表明ProtLGN能够有效挖掘蛋白质定向进化中的正上位效应,为蛋白质的深度进化提供一条有效途径。
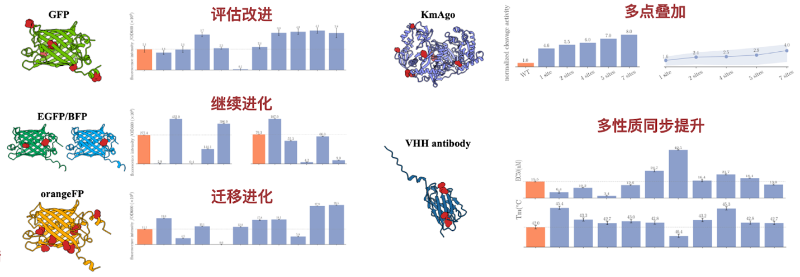
图2 荧光蛋白发光强度(FP),VHH抗体结合强度与热稳定性,以及中温核酸剪切酶(KmAgo)剪切活性的多点位突变结果。
ProtLGN作为一种新型的蛋白质设计工具,为生物学家和药物研发人员提供了一个强大且可靠的计算平台。ProtLGN不仅能够深入解析蛋白质的结构与功能的复杂性,而且能够突破传统蛋白质设计方法遇到的瓶颈,为基于蛋白质的医药研究、生物技术开发等提供了全新并且有效的解决方案。
自然科学研究院/上海国家应用数学中心(上海交通大学分中心)助理研究员周冰心博士,密歇根大学神经科学研究所/细胞与发育生物学研究所博士后郑力荣博士,生命科学技术学院博士研究生吴邦昊,上海人工智能实验室/华东理工大学信息科学与工程学院硕士研究生谭扬为共同第一作者。自然科学研究院/物理与天文学院/张江高等研究院洪亮教授为通讯作者。该工作得到了国家自然科学基金委、上海市科委、教委、上海人工智能国家实验室和张江高等研究院的支持。
论文链接:https://pubs.acs.org/doi/10.1021/acs.jcim.4c00036