
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大赵一雷团队基于人工智能证明生物合成酶预反应态分析的有效性
近日,Cell Press细胞出版社期刊《Cell Reports Physical Science》在线发表了上海交通大学生命科学技术学院赵一雷教授研究团队题为“Understanding the effectiveness of enzyme pre-reaction state by quantum-based machine learning model”的最新成果。生命科学技术学院博士生骆声淦为论文第一作者,上海人工智能实验室刘岚轩等协助了其中机器学习建模工作。该研究以酵母醇脱氢酶CpRCR蛋白质工程中手性醇产物立体选择性预测为应用场景,首先通过传统的伞形采样方法测算了Proleg规则和反Proleg规则下两种预反应态构象的相对稳定性,发现模拟计算结果与实验观察值基本一致。作者进一步采用高阶的量子化学QM/MM方法收集七百余对“预反应态-过渡态”的三维结构信息,利用催化反应中心高达千余的拓扑特征参数展开人工智能机器学习LASSO-SVM回归分析,最后基于前线反应轨道理论解释了为何仅仅数十个精选参数的预反应态QML模型可以有效预测CpRCR酶催化反应活性。
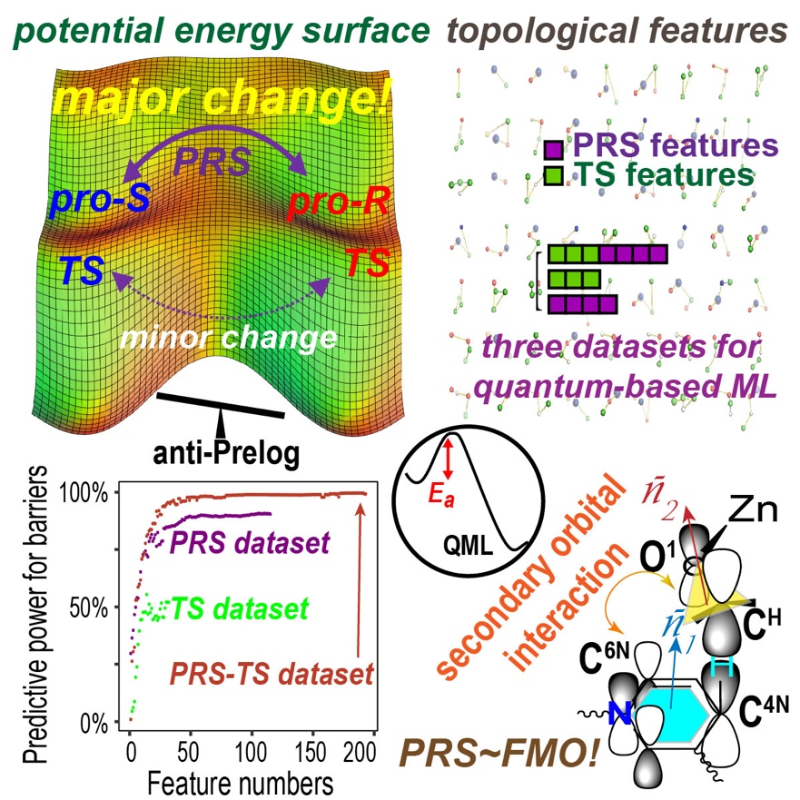
酶催化立体选择性预测在科学工程计算上是一个重大的挑战,它需要在高度复杂大分子系统中用高阶量子力学方法测算不同反应途径之间的微小差别。基于预反应态的快速分子动力学模拟方法已经广泛用于酶催化活性的分析,然而人们在如何选择近进攻构象NAC的预反应态拓扑参数上长期存在各种争议,急需在理论上突破过渡态和预反应态二者对酶催化活性的定量化效应。该课题组基于与江南大学生物工程实验室聂尧课题组长期合作中酶工程科学计算的积累,选用了反应机理清晰可靠、残基突变实验数据丰富、生物工业生产应用广泛的醇脱氢酶CpRCR为研究切入点,利用伞形采样分子动力学预反应态模拟中获得的pro-R和pro-S活性构象,在上海交通大学高性能计算中心展开规模化量子力学计算,通过QM/MM结构数据驱动方法,构建了基于“预反应态-过渡态”联合数据、具有99.6%解释力的高精度人工智能机器学习模型。而后采用相同的建模流程分别构建了基于“预反应态”或“过渡态”独立数据集的两个QML对比模型,发现PRS-QML的酶活预测能力高达90.7%而TS-QML的酶活预测能力只有55.4%,成功地说明了在生物合成酶残基突变和底物改造等实际应用场景中预反应态模型预测具有压倒性的优势。作者进一步利用前线分子轨道理论说明QML模型中所富集到的结构拓扑参数与主-次级轨道相互作用的相关性,阐明了蛋白质工程对酶催化反应势能面的影响主要集中在预反应态区域的变化,而在生物合成酶在天然或人工进化中过渡态区域已经高度优化并相对稳定。
该研究工作为赵一雷课题组长期从事复杂体系反应通路计算系统研究的又一突破性成果,将显著地推进预反应态模型在生物合成酶蛋白质工程改造领域的应用。
该研究获得了国家重点研发计划“合成生物学”重点专项和国家自然科学基金的资助。