
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大欧竑宇研究组发展病原菌耐药移动元件的一键式分析软件
近日,上海交通大学生命科学技术学院、微生物代谢国家重点实验室欧竑宇研究组在分子生物学知名刊物Nucleic Acids Research上发表题为“VRprofile2: detection of antibiotic resistance-associated mobilome in bacterial pathogens”的研究论文。该论文报道了病原菌耐药移动元件一键式分析的新软件VRprofile2,将有助于对多重耐药区形成机制的研究,为遏制细菌耐药性的传播提供新的思路。生命科学技术学院博士生王萌为该论文第一作者,欧竑宇教授为通讯作者。
细菌耐药问题已经成为全球公共健康领域的重大挑战。临床上常见的耐药菌包括Enterococcus faecium (屎肠球菌)、Staphylococcus aureus (金黄色葡萄球菌)、Klebsiella pneumonia (肺炎克雷伯菌)、Acinetobacter baumannii (鲍曼不动杆菌)、Pseudomonas aeruginosa (铜绿假单胞菌)、Enterobacter species (肠杆菌)和Escherichia coli (大肠埃希菌),它们统称为ESKAPEE。这些耐药菌富含种类繁多的可移动遗传元件,主要包括质粒、整合性接合元件、基因组岛、原噬菌体、整合子、转座子和插入序列等。这些移动元件序列约占整个细菌基因组长度的10-30%;该研究组率先提出了用“移动基因组” (mobilome) 来概述细菌移动元件序列的重要性、复杂性和异质性 (Ou, H.Y., et al., Nucleic Acids Research, 2005, 2006, 2007)。在ESKAPEE临床分离株中,接合质粒和整合性接合元件常携带多个获得性耐药基因,而这些耐药基因的两侧常出现插入序列和转座子等其它移动元件,极易发生插入、缺失和重组等遗传事件,进而形成复杂的嵌合结构。
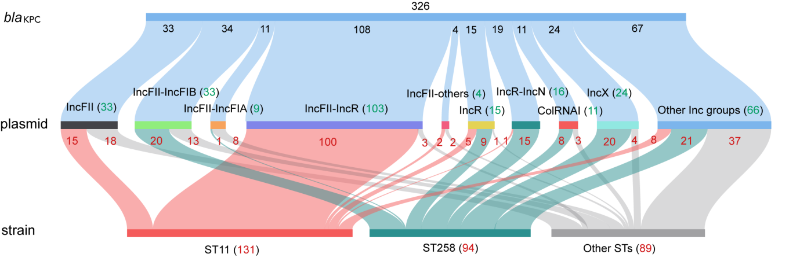
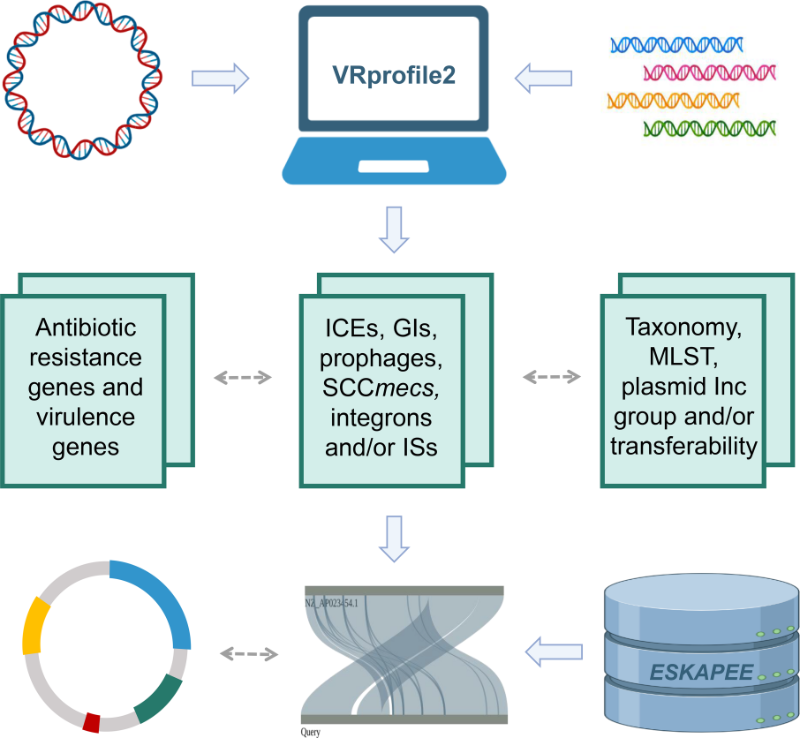
针对ESKAPEE临床株耐药区结构变异的分析需求,VRprofile新版本进行了全新的算法设计和软件实现。在算法设计方面,VRprofile使用“功能模块为主,序列特征为辅”的方法,基于保守基因簇的共定位来识别整合性接合元件等复杂的移动元件。VRprofile它在精准识别不同移动元件的基础上,提出了评估单个细菌中可移动基因组含量的指标mV (mobilome value)。在Web应用方面,VRprofile采用交互式可视化的框架来提供三个特有的分析服务:(i) 一键式识别多种移动元件以及毒力基因和耐药基因,提供了>5500个ESKAPEE细菌移动元件和mV的预计算结果;(ii) 可将识别结果与后台数据库MobilomeDB2收录的一千多个活跃的耐药移动元件进行结构比对,辅助用户解析多重耐药区的结构变异;(iii) 整合病原菌菌株分型和质粒的不相容群和转移潜力等预测功能,为用户探讨“菌型-质粒-耐药基因”的关联性提供便捷的服务,例如下图对碳青霉烯耐药肺炎克雷伯菌的分析。
该项研究得到国家重点研发计划、国家自然科学基金、上海市科委和上海交通大学医工交叉等项目的资助。
论文链接:https://doi.org/10.1093/nar/gkac321