
 探索发现 · 交大智慧
探索发现 · 交大智慧
上海交大欧竑宇研究组开发细菌全基因组规模预测IV型分泌系统效应蛋白的新软件
近日,上海交通大学生命科学技术学院微生物代谢国家重点实验室在生物信息学顶级刊物《Briefings in Bioinformatics》(IF:11.622)上发表题为“T4SEfinder: a bioinformatics tool for genome-scale prediction of bacterial type IV secreted effectors using pre-trained protein language model”的研究论文。该文将人工智能方法应用于细菌IV型分泌系统效应蛋白预测的场景,提供了一款新的、适用于细菌全基因组规模的预测软件。生命科学技术学院硕士生张昱朦和张阳明为该论文的并列第一作者,生命科学技术学院欧竑宇教授和澳大利亚莫纳什大学生物医学发现研究所宋江宁副教授为该论文的共同通讯作者。
细菌IV型分泌系统分泌系统(type IV secretion system, T4SS)是由多个蛋白组件构成的跨膜装置,不仅能够介导DNA接合转移,还能将效应蛋白精准转运到真核或原核细胞中。IV型分泌系统效应蛋白(T4SE)能够影响真核细胞的基因表达和信号转导,在病原细菌致病过程中起到重要作用。前期工作中,欧竑宇研究组构建了T4SS开放数据库SecReT4,通过文本挖掘和人工校正,系统地收录了实验验证的IV型分泌系统基因簇和效应蛋白等高质量数据(Bi, et al., Nucleic Acids Research, 2013)。预测效应蛋白最简便的方法是与SecReT4收录的T4SEs进行序列比对;而人工智能方法则有可能发现与已知T4SE序列相似性低的新效应蛋白。近期,卷积神经网络(CNN)等多种机器学习方法开始应用于T4SE和non-T4SE的分类问题,然而它们的预测精度和运行速度仍有待提高。
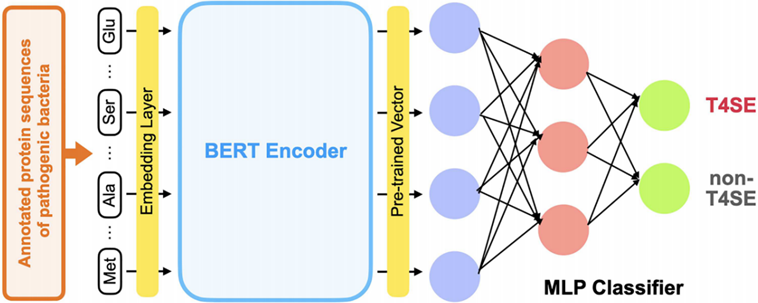
细菌IV型分泌系统效应蛋白预测软件T4SEfinder的模型架构
TAPEBert_MLP主体结构由蛋白序列预训练语言模型(TAPEBert)和多层感知机分类器(MLP)组成
该论文将蛋白序列预训练语言模型(pre-trained protein language model)TAPEBert应用到T4SE分类任务中,开发了快速准确的T4SE预测软件T4SEfinder。首先,通过更新SecReT4数据库,T4SEfinder的训练集包含了来自21种细菌的518个经实验验证的T4SEs。其次,通过综合评估多种机器学习模型对T4SE的预测性能后,T4SEfinder采用了基于BERT的蛋白序列预训练方法TAPE(Task Assessing Protein Embeddings)以及多层感知机分类器(multi-layer perceptron,MLP)来搭建模型TAPEBert_MLP(上图);对于五折交叉验证和独立测试数据集,TAPEBert_MLP模型的预测准确率略优于常用的基于PSSM(位置特异性打分矩阵)和CNN的PSSM_CNN模型。最后,部署在一台RTX 2080 SUPER GPU服务器上的T4SEfinder能够在3分钟内完成5000条蛋白序列中T4SE和non-T4SE的分类,速度比PSSM_CNN方法快五十多倍。因此,T4SEfinder适用于细菌全基因组规模的T4SE识别问题,可能有助于实验发现病原菌的新效应蛋白。
论文链接:https://academic.oup.com/bib/advance-article/doi/10.1093/bib/bbab420/6397152?login=true