
 交大要闻
交大要闻
上海交通大学两支代表队在2023 iGEM国际基因工程机器大赛上摘得一金一银
日前,2023年合成生物学领域国际顶级赛事——国际基因工程机器大赛(International Genetically Engineered Machine Competition,以下简称为iGEM)在法国巴黎落下帷幕,共有来自麻省理工学院、牛津大学、耶鲁大学、清华大学、北京大学、上海交通大学等国内外知名学府的400余支顶尖队伍参赛。上海交通大学代表队SJTU-BioX-Shanghai延续了以往在此项竞赛中的优异表现,凭借项目“The MONCKIE King”蝉联金牌;SJTU-Software凭借项目“DARWINS”荣获“Best Software&AI Project”赛道奖,并获银奖,这也是学校两支队伍近年来首次获得赛道奖,实现了新的突破,再一次展现了交大学子的实力和风采。这也是交大连续第15年摘得该国际顶级赛事的金牌。

SJTU-BioX-Shanghai
SJTU-BioX-Shanghai团队由贺林院士领衔,马钢副研究员、胥川副研究员、王毓舒老师指导,团队成员由来自生命科学技术学院的杨祯熙、江向海、李雨婷、涂然、于子骞、孙浩成、张思瑶、郑雅文,致远学院张可昕、皮家豪、李宣仪,医学院迪拉外尔,生物医学工程学院田钰栋、林成杰,设计学院谢雯妍、郭蓁蓁、戴淑妍、钱思易,共18名本科生组成。2022年团队队长闫奕潇,2021年团队队员朱子健,博士生王晓晴等担任技术顾问。

SJTU-BioX-Shanghai团队将目光投向重金属污染的生物修复。由于历史污染残留或自然污染引起的低浓度重金属污染水体会通过水循环进入人类生产生活,且重金属离子通过食物网的富集作用最终对人体产生毒性。因此,在自然水体中的重金属离子原位去除对人类饮用水和农业渔业生产安全有着至关重要的作用。
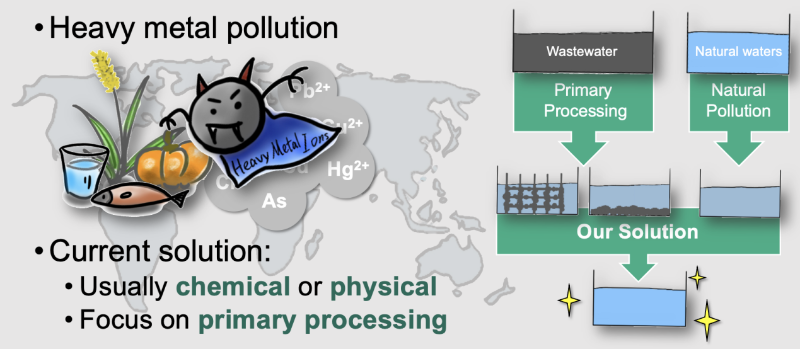
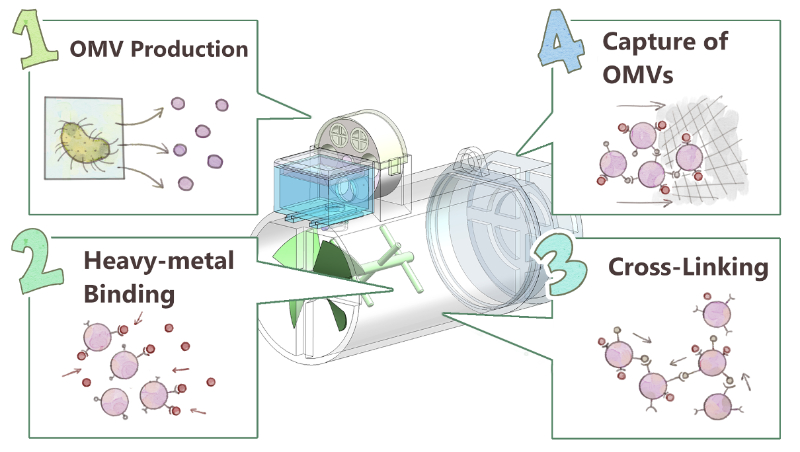
SJTU-BioX-Shanghai创新性地将细菌外膜囊泡(Outer Membrane Vesicles, 后简称OMV)应用在重金属去除。OMV是一种由革兰氏阴性细菌外膜自然产生的脂质囊泡,其组成成分与外膜类似,可用于表面展示目标蛋白。利用外膜锚定蛋白将重金属结合蛋白展示在OMV表面,即可使OMV具备结合重金属离子的功能。

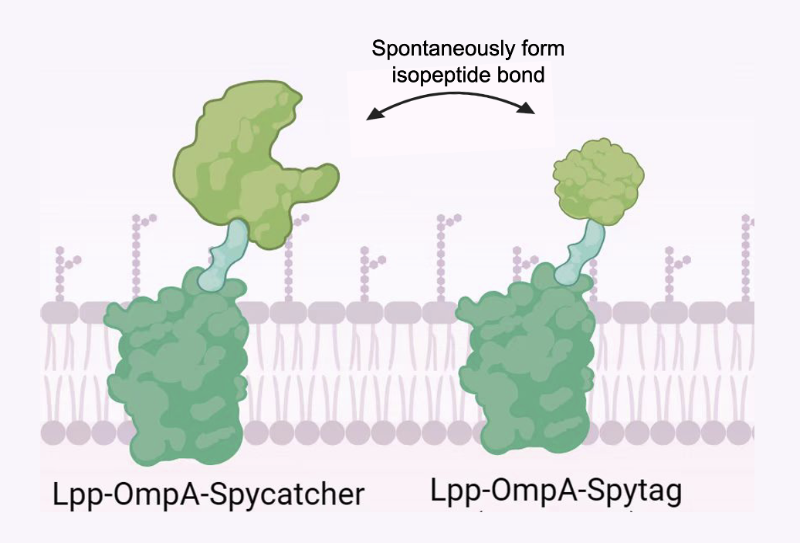
SJTU-BioX-Shanghai设计了使OMV聚集的交联模块。SpyCatcher和SpyTag是一对高效的蛋白接头,两者接近时能自发形成异肽键,从而形成相互交联的蛋白复合体。当仅展示SpyCatcher的OMV和仅展示SpyTag的两种OMV在溶液中混合时,能够相互连接,形成较大体积的聚集体,便于最终的收集与去除。
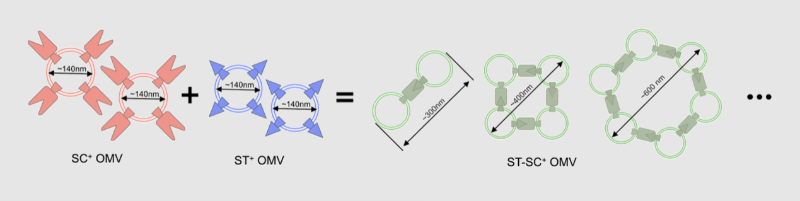
SJTU-BioX-Shanghai还在重金属结合蛋白的外端融合了His-tag,使OMV能够被镍珠或镍网结合,从而完成对OMV聚集体的捕获收集。
在赛季中,SJTU-BioX-Shanghai团队能够顺利完赛,离不开所有队员和老师的辛苦付出,也离不开来自世界各地参赛者的热情帮助。在与多个队伍的相互合作中,参赛队员们收获了友谊,拓宽了视野,留下了精彩的回忆。

SJTU-Software
正值iGEM竞赛20周年,在赛制改革、赛道变迁的背景下,上海交通大学代表队SJTU-Software凭借项目“DARWINS”荣获“Best Software&AI Project”赛道奖,并获银奖。这一优异成绩离不开韦朝春教授和张岳副研究员的指导,同时感谢陈海峰教授、洪亮教授和刘倩副研究员在项目推进过程中提供的宝贵意见以及学校超算中心提供的计算资源。

SJTU-Software团队由韦朝春教授、张岳副研究员担任指导教师,由来自生命科学技术学院的朱骏杰担任队长,成员由魏嘉宁、丁晓涵、钱薪宇、郑睿宁、林雨萱、庄晓熙、沈钰婷、朱乐涛、赵宜辰、杨嘉懿、郑歆瑶、李怡萱、张子骜、黄彦超、贺思齐、徐晨萱共17名本科生组成。
Ago蛋白发挥功能
项目模型
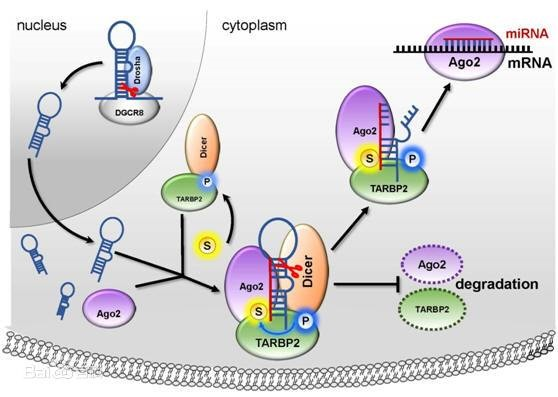
团队的参赛项目为“Darwins: Directed Ago Renewal With Ideal proteiN thermal-Stability”,旨在开发一个蛋白质热稳定性预测平台,根据预测对Ago蛋白进行热稳定性改造,用于检测、诊断等多方面。在文献调研中,团队发现Ago蛋白与传统的CRISPR系统相比,它具有更小巧的体积、更高的敏感性,可以作为一种更为灵活和全面的工具,然而当前大多数Ago蛋白都存在热稳定性欠佳的问题,限制了其在生物技术领域的广泛应用。

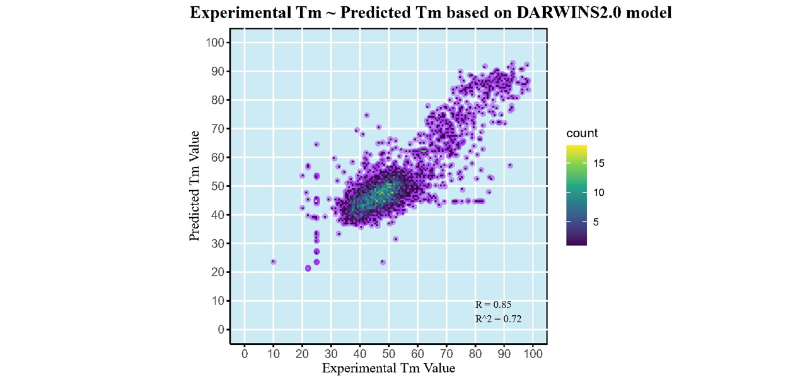
模型拟合效果及预测结果可视化(部分)
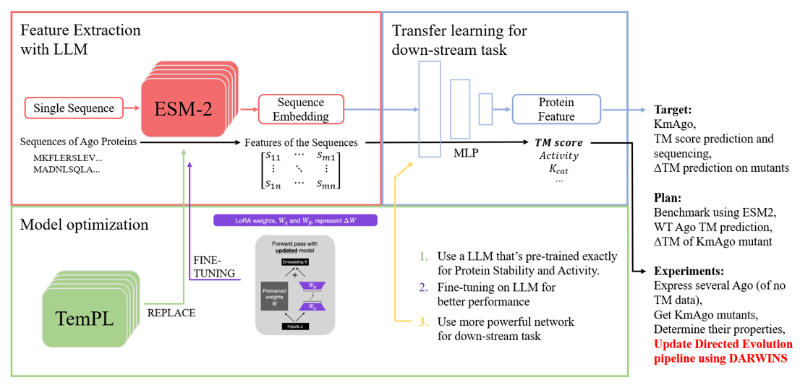
DARWINS平台主要由三个部分组成:蛋白质序列特征提取、下游基于迁移学习的预测、模型优化。团队使用大语言模型ESM2&TempL提取蛋白质的序列特征,并将其编码为特征矩阵作为下游模块的输入,序列特征迁移至下游由迁移学习方法得到的MLP网络进行tm值(即蛋白质构象稳定时对应的温度值)的预测。最后在模型训练的过程中,使用LoRA方法进行模型的微调。最终团队利用该模型辅助KmAgo蛋白的定向进化,获得了更稳定的蛋白序列,表明了该模型的准确性和软件推荐改造方案的可行性。未来会持续进行模型优化和维护,以协助不同领域的研究人员在实验室和其他与Ago蛋白相关的研究。

团队成员支教活动

队员朱乐涛在iGem Grand Jamboree上作项目汇报
与此同时,团队采取多种方式来宣传合成生物学,以及与项目相关的知识。团队在小红书平台上创建了官方账号,让更多人了解合成生物学的重要性和潜在应用,促进科学知识的传播和社会发展;团队也积极走进学校,通过支教活动为中小学生提供近距离探索微观世界的机会,带领学生了解动物和植物的结构,激发更多小朋友对生命科学的兴趣和探索;团队积极与其他高校团队进行交流和合作,包括与交大Bio-X团队和同济大学的合作,参与海南CCiC大会等。
从2009到2023年,上海交通大学iGEM代表队斩获十九金四银二铜,并曾荣获亚洲冠军、最佳人工生物模块设计、全球最佳16队、最佳医学诊断项目、最佳软件项目提名、赛道最佳奖等殊荣。今年,上海交通大学两支代表队远赴法国巴黎参加为期三天的Grand Jamboree。2023届队员秉承“精勤、力行、求真、务实”的奋斗精神,在前沿生物学的世界舞台上再一次捍卫了交大荣誉!